ננוטכנולוגיית DNA
ננוטכנולוגיית DNA הוא תחום מחקר העוסק בעיצוב וייצור מבנים מלאכותיים של חומצות גרעין לשימושים טכנולוגיים. בתחום זה, חומצות גרעין משמשות להנדסת מבנים שאינם ביולוגיים עבור ננוטכנולוגיה ולא כנושאי המידע הגנטי בתאים החיים. שימוש זה מתאפשר על ידי החוקים הנוקשים של זיווג בסיסי חומצות גרעין, אשר מביאים לכך שרק אותם חלקים של גדילי חומצות גרעין עם רצפי בסיסים (אנ') משלימים (אנ') יתחברו יחד כדי ליצור מולקולות חזקות של חומצות גרעין מסוג סליל דו-גדילי (אנ'). הדבר מאפשר תכנון רציונלי (אנ') של רצפי בסיסים אשר יוכלו באופן סלקטיבי ליצור את המבנה הרצוי, המוביל ליכולת ייחודית המאפשרת יצירת מבנים מורכבים וייחודיים בעלי בקרה מדויקת בקנה המידה הננומטרי. DNA הוא החומר הדומיננטי בו משתמשים, אך קיימים מבנים המשלבים חומצות גרעין אחרות, כגון: RNA וחומצות פפטידו-גרעיניות (PNA).
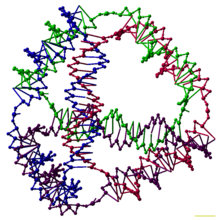
הבסיס הרעיוני של ננוטכנולוגיית DNA נהגה לראשונה על ידי נדריאן סימן (אנ') בראשית שנות ה-80, והתחום החל למשוך התעניינות נרחבת באמצע שנות ה-2000. חוקרים בתחום יצרו מבנים סטטיים כגון מבנים גבישיים דו ותלת־ממדיים, צינורות, פאונים וצורות שרירותיות שנעשו על ידי שיטת ה-DNA אוריגמי. כמו כן הם יצרו מבנים פונקציונליים, כגון מכונות מולקולריות ומחשבי DNA. התחום מתחיל לשמש ככלי לפתרון בעיות במדע בסיסי, בביולוגיה מבנית ובביופיזיקה, כולל יישומי קריסטלוגרפיה וספקטרוסקופיה (אנ') לקביעת מבנה החלבון. פוטנציאל היישומים בתחומי האלקטרוניקה המולקולרית (אנ') והננו-רפואה נחקר אף הוא.
מושגי יסוד עריכה
מאפייני חומצות גרעין עריכה
ננוטכנולוגיה מוגדרת כתחום מחקר של חומרים ומכשירים בעלי תכונות בקנה מידה קטן מ-100 ננומטר. ננוטכנולוגיית DNA וצורות אחרות של הרכבה עצמית מולקולארית מהווים גישה של מטה-מעלה לננוטכנולוגיה, שבה רכיבים מולקולאריים יוצרים באופן ספונטני מבנים מאורגנים ויציבים הודות לתכונות הפיזיקליות והכימיות שלהם.[4] חומצות גרעין, כגון ה-DNA מתאימות מאוד לבנייה בקנה מידה ננומטרי, כמו חומצות גרעין מסוג סליל דו גדילי בעלי קוטר שאורכו 2 ננומטר וסיבוב חוזר של הסליל באורך 3.5 ננומטר. המאפיין המרכזי שהופך חומצות גרעין לשימושיות יותר מחומרים אחרים, לשם בניית מבנים הוא הקישור בין שני גדילי חומצות גרעין שתלוי בחוקים פשוטים של זיווג בסיסים, ויצירת מבנה מסוים על ידי הקישור, כך שקל לשלוט בהרכבת מבני החומצות גרעין דרך עיצוב חומצות גרעין. מאפיין זה אינו חלק ממאפייני חומרים אחרים המשמשים בננוטכנולוגיה, כולל חלבונים, כאשר פעולת עיצוב מבנה החלבון קשה מאוד, וכן ננו-חלקיקים, שהם חסרי יכולת להרכבה ספציפית בכוחות עצמם.[5]
המבנה של מולקולת חומצת גרעין מורכב מרצף נוקליאוטידים, כאשר ההבדל היחיד בינם הוא ברצף הבסיסים. ב־DNA, ארבעת הבסיסים הם: אדנין (A), ציטוזין (C), גואנין (G) ותימין (T). לחומצות גרעין קיימת התכונה ששני בסיסים מתאימים משני גדילים יקשרו זה לזה בקשרי מימן ליצירת סליל דו גדילי כאשר A נקשר רק ל-T ו־C- נקשר רק ל-G.[5][6] מכיוון שזיווג הבסיסים המתאימים הוא תהליך מועדף מבחינה אנרגטית, גדילי חומצות גרעין, במרבית המקרים, יקשרו זה לזה בקונפורמציה הממקסמת את מספר זוגות הבסיסים המותאמים זה לזה. לפיכך, הרצף של הבסיסים בגדילים קובע את דפוס הקשר בין הבסיסים וכן את המבנה הכללי בדרך הניתנת לבקרה בקלות. בננוטכנולוגיית DNA, רצפי הבסיסים בגדילים מעוצבים באופן מתוכנן על ידי חוקרים, כך שהאינטראקציות של זיווגי הבסיסים יגרמו לגדילים להסתדר בקונפורמציה הרצויה.[3][5]
תת תחומים עריכה
תחום הננוטכנולוגיית DNA לעיתים מחולק לשני תת-תחומים חופפים: ננוטכנולוגיית DNA מבנית וננוטכנולוגיית DNA דינמית. ננוטכנולוגיית DNA מבנית (SDN-Structural DNA Nanotechnology), מתמקדת בסינתזה ואפיון קומפלקסים של חומצות גרעין וחומרים המגיעים בסופו של דבר לשיווי-משקל סטטי. מאידך, ננוטכנולוגיית DNA דינמית מתמקדת בקומפלקסים שלא מגיעים לשיווי-משקל, כגון: יכולת הסידור מחדש באמצעות גירוי כימי או פיזי. מספר קומפלקסים, כגון מכשירים ננומכניים של חומצות גרעין, משלבים תכונות של שני התת-תחומים: מבניים ודינמיים.[7][8]
הקומפלקסים שנבנו על ידי ננוטכנולוגיית DNA מבנית משתמשים במבנים מסועפים של חומצות גרעין המכילים צמתים. (לעומת זאת, רוב ה-DNA הביולוגי קיים כסליל דו גדילי לא מסועף). אחד המבנים המסועפים הפשוטים ביותר הוא צומת בעל ארבע זרועות המורכב מארבעה גדילי DNA נפרדים, חלקים מהם משלימים זה את זה בדרך מסוימת. שלא כמו בצומתי Holliday טבעיים, לכל זרוע בצומת המלאכותי קיים |רצף בסיסים שונה, הגורם לנקודת הצומת להיות קבועה במיקום מסוים. צמתים רבים יכולים להשתלב באותו קומפלקס, כמו השימוש הנפוץ במוטיב ה-DX) double-crossover), המכיל שני סלילים דו גדילים מקבילים בעלי גדילים בודדים שחוצים את הסלילים בשתי נקודות חיתוך. כל נקודת חיתוך היא בעצמה צומת בעל ארבע זרועות, אך מוגבלת לכיוון אחד, בניגוד לצומת גמיש יחיד בעל ארבע זרועות, המספק נוקשות שגורמת למוטיב ה-DX להתאים כבלוק לקומפלקסים גדולים יותר של DNA.[3][5] ננוטכנולוגיית DNA דינמית משתמשת במנגנון הנקרא 'מאחז מתווך של עקירת גדיל' המאפשר לקומפלקסים של חומצות הגרעין סידור מחדש בתגובה להוספת גדיל חדש של חומצת גרעין. בתגובה זו, הגדיל הנכנס נקשר לקצה דביק של קומפלקס דו-גדילי, בעקבות כך המערך המועדף מבחינה אנרגטית ישתנה ולכן אחד גדילים הקשורים לקומפלקס המקורי יוסח באמצעות תהליך הגירת הענף.[7] כמו כן, סידור מחדש של מבנים ומכשירים יכול להתבצע באמצעות חומצות גרעין פונקציונליות, כגון דה-אוקסי-ריבוזימים וריבוזימים, אשר מסוגלים לבצע תגובות כימיות בדומה לאנזימים חלבוניים ואפטאמרים אשר יכולים להיקשר לחלבונים ספציפיים או למולקולות קטנות.[9]
היסטוריה עריכה
הבסיס הרעיוני של ננוטכנולוגיית DNA נהגה לראשונה על ידי נדריאן סימן בראשית שנות ה-80.[10] המניע המקורי של סימן היה ליצור גביש DNA תלת־ממדי כדי להתמצא במולקולות גדולות אחרות, כך שמחקר הקריסטלוגרפיה יהפוך לפשוט יותר על ידי הפסקת התהליך הקשה של קבלת גבישים טהורים. הרעיון הגיע אליו, על פי הדיווח, בסוף 1980, לאחר שראה דמיון בין אחד מעבודותיו של האמן הסוריאליסטי M.C. Escher שהתמחה בטכניקת חיתוך עץ לבין מערך צומתי ה-DNA בעלי שש זרועות.[3][11] בעבודה הנקראת 'עומק חיתוך עץ' של Escher מוצגת דמות דמיונית בעלת ארבע זרועות ובתוספת הראש והזנב נראית כמבנה של צומת בעל שש זרועות. מספר מבני DNA מסועפים באופן טבעי היו ידועים באותה עת, כולל שכפול המזלג של ה-DNA וצומת ה־Holliday, אבל התובנה של סימן הייתה שצומתי חומצות גרעין יכולים להיווצר על ידי עיצוב מלאכותי מתאים של רצפי הגדילים, וכי הצמתים הללו יכולים, באופן עקרוני, להיות משולבים כחלק מסריג גבישי נוקשה. המאמר התאורטי הראשון המציע את התוכנית פורסם בשנת 1982, ההדגמה הניסיונית הראשונה של צומת ה-DNA הנייח פורסמה שנה לאחר מכן.[5][12]
ב-1991, פרסמה מעבדתו של סימן את הסינתזה של קובייה העשויה מ־DNA-, הננו-מבנה התלת-ממדי הראשון המיוצר מחומצות גרעין, עבורה קבל את פרס פיינמן בננוטכנולוגיה בשנת 1995. בהמשך היה גם DNA אוקטהדרון קטום. עם זאת, עד מהרה התברר כי מבנים אלה, צורות המצולעים עם צמתים גמישים כמו הקודקודים שלהם, לא היו מספיק נוקשים כדי ליצור סריגים תלת־ממדיים. סימן פיתח מוטיב DX נוקשה יותר, ובשנת 1998, בשיתוף פעולה עם אריק ווינפרי, פרסם את יצירת אריחי DX דו־ממדיים.[3] [[10][69המבנים מבוססי האריח הללו הם בעלי היתרון שהם מספקים את היכולת ליישם מחשבי DNA, שבאו לידי ביטוי על ידי וינפרי ופול רותמונד במאמרם שפורסם ב-2004 על אלגוריתמי הרכבה עצמית של מבנה משולש סירפינסקי, ועליו חלקו את פרס פיינמן בטכנולוגיה בשנת 2006. תובנת המפתח של ווינפרי הייתה שאריחי ה־DX- יכלו לשמש כ[אריחי ואנג], דהיינו ההרכבה שלהם הייתה מסוגלת לייצג חישובים.[10] יצירת הסריג התלת־ממדי פורסמה, לבסוף, על ידי סימן ב-2009, קרוב ל-30 שנה אחרי שהחל להגות אותו.[13]
יכולות חדשות של מבני DNA המשיכו להתגלות לאורך שנות ה-2000. מכונת ה-DNA הראשונה, מוטיב שמשנה את המבנה שלו בתגובה לקלט, הוצגה בשנת 1999 על ידי סימן. מערכת משופרת, שהייתה המכשיר הראשון של חומצות גרעין שעשה שימוש ב"מאחז מתווך של עקירת גדיל", הוצג על ידי ברנרד יורק בשנה שלאחר מכן. ההתקדמות הבאה הייתה ליישם את זה לתנועה מכנית, וב-2004 ו-2005, מספר מערכות DNA נעות הודגמו על ידי קבוצות של סימן, נילס פירס, אנדרו טורברפילד וצ'נגדה מאו.[14] הרעיון של שימוש מערכי DNA לתבנית ההרכבה של מולקולות אחרות כמו ננו-חלקיקים וחלבונים, הוצע לראשונה בשנת 1987 על ידי ברוק רובינסון וסימן,[15] והוצג ב-2006 ו-2007 על ידי הקבוצות של האו יאן, פטר דרוון, ותומאס לבין.[5][16]
בשנת 2006 רותמונד הציג, לראשונה, את טכניקת ה-DNA אוריגמי בשביל ליצור בקלות ובאופן יציב מבני DNA מקופלים באופן שרירותי. רותמונד הציע ששיטה זו תהיה שיטת ביניים בין סריגי ה-DX של סימן, שהשתמשה בגדילים קצרים רבים, וה-DNA אוקטהדרון של ויליאם שי, שכללה בעיקר גדיל אחד ארוך מאוד. ה-DNA אוריגמי של רותמונד מכיל גדיל ארוך אשר הקיפול שלו נעזר בכמה גדילים קצרים. שיטה זו אפשרה יצירת מבנים גדולים יותר מאלו שהיו בעבר, שפחות דורשים טכניקות של עיצוב וסנתוז.[17] DNA אוריגמי היה סיפור השער של מגזין Nature ב-15 במרץ 2006.[18] המחקר של רותמונד הציג מבנים דו-ממדיים של ה-DNA אוריגמי בעקבות הדגמה של DNA אוריגמי מוצק תלת-ממדי שהוצג ב-2009 על ידי דאגלס,[19] בעוד המעבדות של יורגן קמס ויאן הציגו מבנים תלת־ממדיים חלולים שנעשו ממבנים דו-ממדיים.[13]
ננוטכנולוגיית DNA נתקלה בתחילה בסקפטיות מסוימת עקב השימוש הלא ביולוגי החריג של חומצות גרעין כחומרים המשמשים לבניית מבנים וכמייצגים תהליכי חישוב. מאמרו של סימן ב-1991 על סינתזה של קוביית DNA נדחתה על ידי כתב העת Science לאחר שמבקר אחד שיבח את המקוריות שלו, בעוד אחר מתח ביקורת על היעדר קשר ביולוגי. עם זאת בתחילת 2010 התחום החל להגדיל את היכולות שלו עד כדי כך שיישומים במחקר בסיסי החלו לבוא לידי ביטוי ויישומים מעשיים ברפואה ובתחומים אחרים החלו להיחשב לריאליים.[13][20]התחום גדל ממספר מועט של מעבדות שעסקו בננוטכנולוגיית DNA בשנת 2001 למעל ל-60 בשנת 2010, תהליך שקידם מדעית את תחום זה בעשור הראשון של המאה ה-21.[21]
חומרים ושיטות עריכה
רצפי הגדילים הבודדים של DNA המרכיבים את המבנה הסופי תוכננו באמצעות הדמיה מולקולארית ותוכנה המדמה את המודל התרמודינמי.[22][23] לאחר תכנון הרצפים, הם סונתזו על ידי השיטות הרגילות ליצירת אוליגו-נוקלאוטידים. תהליך זה הוא אוטומטי בדרך כלל באמצעות מכונה שנקראת oligonucleotide synthesizer, וחומצות גרעין של רצף מותאם אישית, הזמינים מסחרית על ידי ספקים רבים.[24] עבור שיטות הדורשות גדילים טבעיים, טהורים בריכוז ידוע, יש צורך לנקות את הגדילים באמצעות דנטורציה על ידי ג'ל אלקטרופורזה.[25] הריכוזים נקבעים באמצעות אחת מכמה שיטות המכמתות חומצות גרעין, אחת מהן היא השיטה הספקטרוסקופית בה מודדים את בליעת אורכי הגל בתחום האולטרה סגול המאפיינים את ספקטרום הבליעה של רצפי הנוקליאוטידים.[26]
מבני היעד הסופיים מאופיינים בדרך כלל על ידי ג'ל אלקטרופורזה מתאים, אשר מספק מידע על גודל וצורת הקומפלקסים של חומצות הגרעין. שיטת EMSA יכולה לציין אם המבנה כולל את כל הגדילים הרצויים.[27] בתיוג פלואורסצנטי וFRET משמשים לעיתים לאפיון המבנה והקומפלקסים.[28]
ניתן לדמות מבני חומצות גרעין על ידי מיקרוסקופ כוח אטומי, המתאים למבנים ארוכים דו-ממדיים, אך הוא פחות שימושי עבור מבנים תלת־ממדיים עקב האינטראקציה של טיפ המיקרוסקופ עם מבנה חומצות הגרעין השבריריות. עבור הדמיה תלת־ממדית, מיקרוסקופ אלקטרונים חודר ,TEM, וקריו מיקרוסקופ אלקטרונים- cryo-EM הן שיטות חשובות. סריגים תלת־ממדיים מורחבים מנותחים על ידי קריסטלוגרפיה בקרני רנטגן.[29][30]
עיצוב עריכה
ננו-מבני DNA חייבים להיות מעוצבים באופן רציונלי, כך שגדילים בודדים של חומצות גרעין ירכיבו את המבנים הרצויים. תהליך זה מתחיל בדרך כלל עם מפרט של מבנה היעד הרצוי או הפונקציונליות שלו. לאחר מכן נקבע המבנה השניוני הכולל של הקומפלקס, תוך פירוט סידור הגדילים של החומצות גרעין בתוך המבנה והחלקים מהגדילים הללו שצריכים להקשר זה לזה. השלב האחרון הוא תכנון המבנה הראשוני, המהווה את פירוט רצפי הבסיסים בפועל של כל גדיל חומצות גרעין.[31][22]
עיצוב מבני עריכה
הצעד הראשון בעיצוב ננו-מבני חומצות גרעין הוא להחליט כיצד מבנה נתון צריך להיות מיוצג על ידי סידור מסוים של גדילי חומצות גרעין. שלב זה קובע את המבנה השניוני או את עמדותיהם של זוגות בסיסים אשר מחזיקים את הגדילים בודדים יחד לצורה הרצויה.[31] מספר שיטות הוצגו:
• מבנים מבוססי אריחים. גישה זו מחלקת את המבנה הרצוי הסופי לתת יחידות קטנות יותר בעלות קשר חזק בין הגדילים בכל תת-יחידה ואינטראקציות חלשות יותר בין התת-יחידות. משתמשים בשיטה זו, לעיתים קרובות, כדי ליצור, באופן זמני, סריגים בעלי מבנה מחזורי, אך יכול לשמש גם כדי ליישם אלגוריתמית הרכבה עצמית של מבנה. דבר זה הופך אותם לפלטפורמה לחישוב על ידי DNA. שיטה זו הייתה השיטה הדומיננטית מאמצע שנות ה-90 עד אמצע שנות ה-2000, כאשר שיטת ה-DNA אוריגמי התפתחה.[31][32]
• קיפול מבנים. אלטרנטיבות לשיטה הקודמת הן שיטות קיפול, בהן גדיל ארוך יחיד מתקפל לננו-מבנה. הגדיל הארוך יכול להיות רצף מתוכנן המתקפל לפי האינטראקציות שלו עם עצמו, או שזה יכול להתקפל לצורה הרצויה על ידי שימוש בגדילים קצרים יותר. שיטה נוספת נקראת DNA אוריגמי, המאפשרת יצירת צורות דו ותלת-ממדיות בקנה מידה ננומטרי. (ראה מבנים בדידים להלן).[12][18]
• הרכבה דינאמית. שיטה זו שולטת באופן ישיר בקינטיקה של ההרכבה העצמית של ה-DNA, תוך פירוט כל חומרי הביניים של מנגנון התגובה נוסף על התוצר הסופי. הדבר נעשה באמצעות מבנה stem-loop של חומצות הגרעין בחומר בתחילת התגובה, אשר מסתדר לאחר קסקדת התגובה בסדר מסוים. לשיטות הללו יש את היתרון של הליך איזותרמי, ולכן לא כולל את שלב ה-annealing, הנדרש על ידי גישות תרמודינמיות בלבד.[12][33]
עיצוב רצפי עריכה
אחרי כל השיטות הנ"ל, המשמשות לתכנון המבנה השניוני של קומפלקס מסוים יש צורך לתכנן את רצף הנוקליאוטידים שייצור את המבנה הרצוי. בתכנון מקצים רצף בסיסי חומצות גרעין לכל אחד מהגדילים המרכיבים את המבנה כך שהם יצרו את הקונפורמציה הרצויה. הצד השווה בכל שיטות התכנון הוא המטרה שהמבנה הסופי המתוכנן יהיה בעל האנרגיה הנמוכה ביותר מבין כל המבנים האפשריים, ולכן הוא יועדף מבחינה תרמודינמית. הדבר נעשה הן באמצעות שיטות [היוריסטיקה|יוריסטיות] פשוטות ומהירות, כגון מזעור הסימטריה ברצף המאפשרת קיפול הגדיל על עצמו או על ידי שימוש במודל התרמודינמי nearest-neighbor, שהוא מדויק יותר אך איטי יותר ואף יותר אינטנסיבי מבחינה חישובית. מודלים גאומטריים משמשים לבחינת מבנה שלישוני של הננו-מבנים ועל מנת להבטיח שהקומפלקסים לא מתוחים יתר על המידה.[22][23]
עיצוב חומצות גרעין דומה לעיצוב חלבונים. בשניהם רצף של מונומרים מתוכנן כך שהוא יעדיף את המבנה הרצוי ולא יעדיף אף מבנה אחר. לעיצוב חומצות גרעין יש יתרון על עיצוב חלבון בשל כך שהוא קל יותר מבחינה התכנון, כי חוקי הזיווג בסיסים מספיקים כדי לחזות את המבנה המועדף, ואין צורך במידע מפורט על קיפול תלת־ממדי של המבנה. דבר זה מאפשר שימוש בשיטות יוריסטיות פשוטות, שמניבות עיצובים יציבים מבחינה ניסויית. עם זאת, מבני חומצות גרעין הם פחות מגוונים מבחינה פונקציונלית מהחלבונים בגלל היכולת של חלבונים להתקפל למבנים מורכבים, כמו גם מגוון כימי מוגבל של ארבעת הנוקליאוטידים, לעומת 20 חומצות אמינו המרכיבות חלבון.[23]
ננוטכנולוגיית DNA מבנית עריכה
ננוטכנולוגיית DNA מבנית, נקראת באופן מקוצר Structural DNA Nanotechnology -SDN, מתמקדת בסינתזת ואפיון קומפלקסים של חומצות גרעין. לסליל הכפול של חומצות הגרעין יש גאומטריה תלת־ממדית יציבה ומוגדרת המאפשרת לחזות ולתכנן את המבנים של קומפלקסים מורכבים יותר של חומצות גרעין. מבנים רבים כאלה נוצרו, כולל מבנים דו ותלת-ממדיים, מחזוריים, לא מחזוריים ומבנים בדידים.[8]
סריגים מורחבים עריכה
קומפלקסים קטנים של חומצות גרעין יכולים להיות בעלי קצוות דביקים ומשולבים בסריגים דו-ממדיים גדולים יותר, המכילים דפוס מסוים של פסיפס מהאריחים המולקולאריים הבודדים.[8] הדוגמה הראשונה לזה הייתה קומפלקסי ה־DX- ששמשו כאריחי הבסיס, המכילים כל אחד ארבעה קצוות דביקים עם רצפים שגרמו ליחידות ה-DX ליצור יחד יריעות שטוחות דו-ממדיות שהם בעצם גביש קשיח דו־ממדי של DNA.[34][35]מערכים דו-ממדיים נוצרו מתוך מוטיבים שונים, כולל סריג מעויני צומת Holliday,[36] ומגוון מערכים המבוססים על DX.[37][38]
ניתן להציג על ידי מערכים דו-ממדיים מבנים לא מחזוריים אשר הרכבתם מיישמת אלגוריתם לוגי או חישובי.[21] אריחי ה-DX יכולים להכיל רצפי קצוות דביקים נבחרים, כך שהם משמשים כאריחי ואנג, המאפשרים להם לבצע חישוב. הוצג מערך DX שהרכבתו מקודדת לפעולה ; XOR זה מאפשר למערך ה-DNA ליישם את האוטומט התאי שיוצר פרקטל המכונה Sierpinski gasket.[39] למערכת אחרת יש את הפונקציה של מונה בינארי, המציגה מספר שגדל בינארית ככל שהיא גדלה. תוצאות אלו מראות כי החישוב יכול להיות משולב בתוך מכלול של מערכי ה-DNA.[40]
מערכי DX נעשו כדי ליצור צינורות חלולים בקוטר 4–20 ננומטר, שהם סריגים דו־ממדיים שמסתובבים סביב עצמם.[31] צינורות ה־DNA הללו דומים במקצת בגודלם ובצורתם לננו צינורות פחמן. לעומת ננו צינורות פחמן, צינוריות ה־DNA משתנים בקלות רבה יותר ומתחברים למבנים אחרים. אחת הדרכים לבניית צינורות ה־DNA משתמשת בסריג של אריחי DX מעוקלים שמסתובבים סביב עצמו ונסגרים לידי צינור.[43] יצירת סריגים תלת־ממדיים של DNA הייתה המטרה הראשונה בננוטכנולוגיית DNA, אך התברר שזה אחד הדברים הקשים להבנה. הצלחה, באמצעות השימוש במוטיב, מבוססת על רעיון של טנסגריטי, איזון בין מתח ודחיסת כוחות, דווח לבסוף ב-2009.[21][44]
מבנים בדידים עריכה
חוקרים סנתזו מספר קומפלקסים תלת־ממדיים של DNA במבנה של פאונים, כמו קובייה או אוקטהדרון.[12] ההצגות הראשונות של ה-DNA רבפאון היו לאחר עבודה מאוד אינטנסיבית, הדורשת DNA ליגאז ושלבים של סינתזת פאזת מוצק כדי ליצור רבפאון catenated.[45] עבודה רצופה הניבה רבפאון שהסינתזה שלו הייתה הרבה יותר קלה. היא כללה DNA אוקטהדרון עשוי מגדיל אחד ארוך שנועד להתקפל לקונפורמציה נכונה,[46] וטטרהדרון שניתן להפיק מארבעה גדילי DNA בצעד אחד, כפי שמתואר בראש מאמר זה.[1]
ננו-מבנים שרירותיים, בעלי צורות לא רגילות עשויים בדרך כלל בשיטת ה-DNA אוריגמי. מבנים אלה כוללים גדיל ארוך וטבעי של נגיף, שעשוי להתקפל לצורה הרצויה על ידי גדילים בסיסיים קצרים שנועדו לצורך חישוב. לשיטה זו יש את היתרונות של הקלות בעיצוב, כמו רצף הבסיסים שנקבע מראש בגדיל הארוך, ולא דורש ניקיון גבוה של הגדיל וסטויכיומטריה מדויקת, כמו שרוב שיטות ננוטכנולוגיית DNA האחרות עושות. DNA אוריגמי הוצג לראשונה עבור צורות דו־ממדיות, כמו סמיילי ומפה מחוספסת של צפון אמריקה.[12][18] מבנים מוצקים תלת־ממדיים יכולים להיווצר באמצעות סלילי DNA מקבילים מסודרים בתבנית חלת דבש,[19] ומבנים עם מעטפת דו־ממדית שניתן לקפל לתוך צורה חלולה תלת־ממדית, הדומה לקופסת קרטון. אלה יכולים להיות מתוכננים לפתוח ולחשוף או לשחרר מטען מולקולרי בתגובה לגירוי, מה שהופך אותם לפוטנציאליים כמו כלובים מולקולריים מתוכננים.[47][48]
ייצור בתבניות עריכה
במבני חומצות גרעין ניתן לשלב מולקולות אחרות מאשר חומצות גרעין, שלעיתים מכונות "הטרואלמנטים", הכוללים חלבונים, ננו-חלקיקים מתכתיים, נקודות קוונטיות, ופולרנים. דבר זה מאפשר בנייה של חומרים ומכשירים עם מגוון פונקציות גדול יותר משל חומצות גרעין בלבד. המטרה היא להשתמש בהרכבה עצמית של מבני חומצות גרעין לתבנית אשר בה הננו-חלקיקים "מתארחים" בהם, ושולטים במיקומם ואף בכיוונם של האלמנטים.[12][16] רבים מהמערכות האלה השתמשו בקשר קוולנטי, באמצעות אוליגונוקלאוטידים עם אמיד או תיול המשמשים כעין נקודת אחיזה כימית כדי לחבר את ההטרואלמנטים. שיטת זו המשתמשת בקשרים קוולנטיים שמשה כדי לארגן ננו-חלקיקי זהב על מערך DX,[49] וכן לארגן חלבון סטרפטאבידין בתבניות מסוימות במערך DX.[50] מערכת מארחת שאינה קוולנטית המיושמת באמצעות פוליאמידי Dervan על מערך DX שימשה לארגון חלבוני סטרפטאבידין בתבנית ספציפית על מערך DX.[51] ננו-צינורות פחמן המתארחים על מערכי DNA בתבנית המאפשרת הרכבה, משמשים ליצירת טרנזיסטור אלקטרוני מולקולרי, הנקרא carbon nanotube field-effect transistor.[52] בנוסף, ישנן שיטות בהן חומצת גרעין מוחלפת במתכת המחליפה את הצורה הכללית של מבנה חומצת הגרעין המקורית,[53] וכן מתוכנן שימוש בננו-מבני חומצות גרעין כדי ליישם מסכות ליתוגרפיה מולקולרית המאפשרות את העברת התבנית של המבנים אל משטח מוצק.[54]
ננוטכנולוגיית DNA דינאמית עריכה
ננוטכנולוגיית DNA דינאמית מתמקדת ביצירת מערכות חומצות גרעין בעלות פונקציונליות דינאמית הקשורה למבנה שלהם, כגון יצירת מודלים חישוביים ואלגוריתמיים ותנועה מכנית. קיימת חפיפה בין ננוטכנולוגיה DNA מבנית ודינאמית, כמו מבנים שיכולים להיווצר באמצעות annealing ואז יסתדרו מחדש באופן דינמי, או שניתן ליצור אותם באופן דינאמי מלכתחילה.[12][14]
מכשירים ננומכניים עריכה
קומפלקסי ה-DNA הללו משנים את הקונפורמציה שלהם בעקבות גירוי כלשהו מה שהופך אותם לסוג של ננורובוטים. מבנים אלה נוצרים תחילה באופן זהה למבנים סטטיים שנעשו על ידי ננוטכנולוגיית DNA מבנית, אלא שהם מתוכננים כך שיוכלו לשנות קונפורמציה באופן דינמי לאחר הסידור הראשוני.[7][14] המכשיר הראשון מסוג זה עשה שימוש במעבר בין צורות B-DNA ו-Z-DNA על ידי תנועת פיתול כתגובה לשינוי בתנאי הבופר.[55] מערכות נוספות המשנות מצב קיים מתבססות על נוכחותם של גדילי בקרה, המאפשרים למספר מכשירים לפעול באופן עצמאי בתמיסה. כמה דוגמאות של מערכות כאלה הן "פינצטות מולקולאריות" שבהן יש מצב פתוח וסגור,[56] מכשיר שיכול לעבור מקונפורמית PX לקונפורמציית JK2, תוך כדי שימוש בתנועה סיבובית,[57] ומערך דו-ממדי שיכול באופן דינמי להתרחב ולהתכווץ בתגובה לגדילי הבקרה.[58] מבנים דינמיים בהם יש מצב פתוח וסגור, הם בעלי פוטנציאל להתנהג כמו כלוב מולקולרי, כלומר לשחרר או לחשוף את המטען הפונקציונלי בעת הפתיחה.[47][59][60]
הליכוני DNA הם קבוצת ננו-מכונות של חומצות גרעין שמופיעה בהן תנועה כיוונית לאורך מסלול ליניארי. מספר רב של תוכניות הוצגו:[14] אסטרטגיה אחת היא לשלוט בתנועת ההליכון לאורך המסלול באמצעות גדילי בקרה שצריך להוסיף באופן ידני לרצף.[61][62] גישה אחרת היא להשתמש באנזימי רסטריקציה או בדה-אוקסי-ריבוזימים כדי להדביק את הגדילים ולגרום להליכון לנוע קדימה, וכך יש לו את היתרון שהוא פועל באופן עצמאי.[63][64] מערכת שהופיעה מאוחר יותר יכולה ללכת על משטח דו־ממדי ולא במסלול ליניארי, ומציגה את היכולת להרים ולהזיז מטען מולקולרי באופן סלקטיבי.[65] בנוסף, הוצג הליכון ליניארי שמבצע סינתזת DNA בתבניות ככל שמתקדם לאורך המסלול, וכך מאפשר ביצוע שלבים רבים בסינתזה כימית ישירות על ידי ההליכון.[66]
קסקדות עקירת גדילים עריכה
תגובות של קסקדות עקירת גדילים יכולות לשמש עבור מטרות חישוביות או מבניות. תגובה אחת של עקירת גדיל כרוכה בחשיפת רצף חדש בתגובה לנוכחות של חלק מגדיל אחר. תגובות רבות מסוג זה ניתן לקשר לקסקדה שבה כאשר נחשף רצף פלט חדש מתגובה אחת ניתן ליצור תגובה אחרת של עקירת גדיל במקום אחר. זה בתורו מאפשר בניית רשתות של תגובות כימיות עם מרכיבים רבים, המציגות יכולות של חישובים מורכבים ועיבוד אינפורמציה. הקסקדות האלה נעשות תחת העדפה אנרגטית דרך היווצרות של זוגות בסיסים חדשים או רווח האנטרופיה הנובע מתגובות פירוק. קסקדות עקירת גדילים מאפשר פעולה איזותרמית של ההרכבה או תהליך חישובי, לעומת דרישת הרכבת חומצת גרעין המסורתית לשלב annealing תרמי, שבו הטמפרטורה עולה ואז לאט יורדת כדי להבטיח היווצרות נכונה של המבנה הרצוי.[7][33]
עקירת קומפלקסים יכולה לשמש להכנת שערים לוגיים מולקולאריים המסוגלים לבצע חישוב מורכב. שלא כמו במחשבים אלקטרוניים מסורתיים, המשתמשים בכניסות ויציאות של זרם חשמלי, מחשבים מולקולאריים משתמשים בריכוזים של חומרים כימיים ספציפיים כאותות. במקרה של מעגלי עקירת חומצות גרעין, האות הוא נוכחותם של גדילי חומצות גרעין שנצרכו או שוחררו על ידי אירועי קישור או חוסר קישור לגדילים אחרים. בגישה זו נעשה שימוש כדי ליצור שערים לוגיים כגון שערי AND, OR ו־NOT.[67] לאחרונה, הוכח שע"י מעגל ארבע סיביות ניתן לחשב את השורש הריבועי של מספרים שלמים בטווח 0–15, באמצעות מערכת שערים המכילה 130 גדילי DNA.[68]
שימוש נוסף של קסקדות עקירת גדילים הוא בהרכבת מבנים באופן דינמי. אלה משתמשים במבנה stem-loop של המגיבים, כך שכאשר נקשר גדיל קלט, הרצף החדש שנחשף מאפשר להוסיף stem-loops חדשים לקומפלקס שהולך וגדל. בגישה זו נעשה שימוש כדי ליצור מבנים פשוטים כגון צמתים בעלי 3–4 זרועות ודנדימרים.[33]
יישומים עריכה
ננוטכנולוגיית DNA היא אחת הדרכים היחידות ליצירת מבנים מורכבים ובעלי מטרה, בעלי שליטה מדויקת בקנה מידה ננומטרי. בשנים האחרונות מתחילים לראות בתחום זה יישומים לפתרון בעיות במדע בסיסי בביולוגיה מבנית וביופיזיקה. יישום אחד כזה, המוקדם ביותר בתחום, הוא בקריסטלוגרפיה, שבה מולקולות שקשה לגבש בנפרד יכולות להיות מסודרות בסריג תלת־ממדי של חומצות גרעין, ובכך מאפשר את הצגת המבנה שלהם. יישום אחר הוא שימוש במקלות של DNA אוריגמי כדי להחליף גבישים נוזליים בניסויים מסוג residual dipolar coupling ב-NMR חלבוני. הליכוני DNA שימשו כקווי ייצור בקנה מידה ננומטרי כדי להזיז ננו חלקיקים ולשם סינתזה כימית ישירה. יתר על כן, מבני ה-DNA אוריגמי סייעו למחקרים ביופיזיקליים של פונקציית אנזים וקיפול חלבונים.[8][13]
ננוטכנולוגיית DNA מתקדמת לכיוון יישומים פוטנציאליים בעולם האמיתי. היכולת של מערכי חומצות גרעין לסדר מולקולות אחרות מצביע על יישומים אפשריים שלה בתחום האלקטרוניקה בקנה מידה מולקולרי. הרכבת מבנה חומצות גרעין יכול לשמש כתבנית הרכבה של כמה אלמנטים אלקטרוניים מולקולריים כמו ננוחוטים, המספקים שיטה לבקרה בקנה מידה ננומטרי של המיקום והאדריכלות הכוללת של מכשיר אנלוגי לbreadboard מולקולרי.[8][12] ננוטכנולוגיית DNA הושוותה לרעיון של חומר הניתן לתכנות בגלל הוספת יכולת החישוב לתכונות החומר שלו.[69]
ישנם יישומים פוטנציאליים עבור ננוטכנולוגיית DNA בננורפואה, על ידי שימוש ביכולתה לבצע חישוב במצב של תאימות ביולוגית וכך ליצור "תרופות חכמות" עבור תהליכי שיגור תרופות ממוקד. מערכת אחת כזו משתמשת בקופסת DNA חלולה המכילה חלבונים שגורמים לאפופטוזה, או מוות של תאים. קופסית זו תפתח כאשר תהיה בקרבת תא סרטני ותרוקן את החלבון הקטלני.[13][70] יש עניין נוסף ליישם את המבנים המלאכותיים האלו בתאים חיים מהונדסים של חיידקים.
ראו גם עריכה
קישורים חיצוניים עריכה
הערות שוליים עריכה
- ^ 1 2 DNA polyhedra: Goodman, Russel P.; Schaap, Iwan A. T.; Tardin, C. F.; Erben, Christof M.; Berry, Richard M.; Schmidt, C.F.; Turberfield, Andrew J. (9 December 2005). "Rapid chiral assembly of rigid DNA building blocks for molecular nanofabrication". Science 310 (5754): 1661–1665. Bibcode 2005Sci...310.1661G. doi:10.1126/science.1120367. PMID 16339440
- ^ 1 2 3 Overview: Mao, Chengde (December 2004). "The emergence of complexity: lessons from DNA". PLoS Biology 2 (12): 2036–2038. doi:10.1371/journal.pbio.0020431. PMC 535573. PMID 15597116. ^ a b c d e Overview: Seeman, Nadrian C. (June 2004). "Nanotechnology and the double helix". Scientific American 290 (6): 64–75. doi:10.1038/scientificamerican0604-64. PMID 15195395.
- ^ 1 2 3 4 5 Overview: Seeman, Nadrian C. (June 2004). "Nanotechnology and the double helix". Scientific American 290 (6): 64–75. doi:10.1038/scientificamerican0604-64. PMID 15195395.
- ^ Background: Pelesko, John A. (2007). Self-assembly: the science of things that put themselves together. New York: Chapman & Hall/CRC. pp. 5, 7. ISBN 978-1-58488-687-7.
- ^ 1 2 3 4 5 6 Overview: Seeman, Nadrian C. (2010). "Nanomaterials based on DNA". Annual Review of Biochemistry 79: 65–87. doi:10.1146/annurev-biochem-060308-102244. PMID 20222824.
- ^ Long, Eric C. (1996). "Fundamentals of nucleic acids". In Hecht, Sidney M. Bioorganic chemistry: nucleic acids. New York: Oxford University Press. pp. 4–10. ISBN 0-19-508467-5.
- ^ 1 2 3 4 Dynamic DNA nanotechnology: Zhang, David Y.; Seelig, Georg (February 2011). "Dynamic DNA nanotechnology using strand-displacement reactions". Nature Chemistry 3 (2): 103–113. doi:10.1038/nchem.957. PMID 21258382
- ^ 1 2 3 4 5 Structural DNA nanotechnology: Seeman, Nadrian C. (November 2007). "An overview of structural DNA nanotechnology". Molecular Biotechnology 37 (3): 246–257. doi:10.1007/s12033-007-0059-4. PMID 17952671
- ^ Dynamic DNA nanotechnology: Lu, Yi; Liu, Juewen (December 2006). "Functional DNA nanotechnology: Emerging applications of DNAzymes and aptamers". Current Opinion in Biotechnology 17 (6): 580–588. doi:10.1016/j.copbio.2006.10.004. PMID 17056247
- ^ 1 2 3 History: Pelesko, John A. (2007). Self-assembly: the science of things that put themselves together. New York: Chapman & Hall/CRC. pp. 201, 242, 259. ISBN 978-1-58488-687-7.
- ^ History: See "Current crystallization protocol". Nadrian Seeman Lab. for a statement of the problem, and "DNA cages containing oriented guests". Nadiran Seeman Laboratory. for the proposed solution.
- ^ 1 2 3 4 5 6 7 8 Overview: Pinheiro, Andre V.; Han, Dongran; Shih, William M.; Yan, Hao (December 2011). "Challenges and opportunities for structural DNA nanotechnology". Nature Nanotechnology 6 (12): 763–772. doi:10.1038/nnano.2011.187. PMID 22056726.
- ^ 1 2 3 4 5 History/applications: Service, Robert F. (3 June 2011). "DNA nanotechnology grows up". Science 332 (6034): 1140–1143. doi:10.1126/science.332.6034.1140.
- ^ 1 2 3 4 DNA machines: Bath, Jonathan; Turberfield, Andrew J. (May 2007). "DNA nanomachines". Nature Nanotechnology 2 (5): 275–284. Bibcode 2007NatNa...2..275B. doi:10.1038/nnano.2007.104. PMID 18654284.
- ^ Nanoarchitecture: Robinson, Bruche H.; Seeman, Nadrian C. (August 1987). "The design of a biochip: a self-assembling molecular-scale memory device". Protein Engineering 1 (4): 295–300. doi:10.1093/protein/1.4.295. PMID 3508280.
- ^ 1 2 Overview: Endo, Masayuki; Sugiyama, Hiroshi (12 October 2009). "Chemical approaches to DNA nanotechnology". ChemBioChem 10 (15): 2420–2443. doi:10.1002/cbic.200900286. PMID 19714700.
- ^ DNA origami: Rothemund, Paul W. K. (2006). "Scaffolded DNA origami: from generalized multicrossovers to polygonal networks". In Chen, Junghuei; Jonoska, Natasha; Rozenberg, Grzegorz. Nanotechnology: science and computation. Natural Computing Series. New York: Springer. pp. 3–21. doi:10.1007/3-540-30296-4_1. ISBN 978-3-540-30295-7.
- ^ 1 2 3 DNA origami: Rothemund, Paul W. K. (16 March 2006). "Folding DNA to create nanoscale shapes and patterns". Nature 440 (7082): 297–302. Bibcode 2006Natur.440..297R. doi:10.1038/nature04586. PMID 16541064.
- ^ 1 2 DNA origami: Douglas, Shawn M.; Dietz, Hendrik; Liedl, Tim; H?gberg, Bj?rn; Graf, Franziska; Shih, William M. (21 May 2009). "Self-assembly of DNA into nanoscale three-dimensional shapes". Nature 459 (7245): 414–418. Bibcode 2009Natur.459..414D. doi:10.1038/nature08016. PMC 2688462. PMID 19458720.
- ^ History: Hopkin, Karen (August 2011). "Profile: 3-D seer". The Scientist. Retrieved 8 August 2011.
- ^ 1 2 3 History: Seeman, Nadrian (9 June 2010). "Structural DNA nanotechnology: growing along with Nano Letters". Nano Letters 10 (6): 1971–1978. Bibcode 2010NanoL..10.1971S. doi:10.1021/nl101262u. PMC 2901229. PMID 20486672.
- ^ 1 2 3 Design: Brenneman, Arwen; Condon, Anne (25 September 2002). "Strand design for biomolecular computation". Theoretical Computer Science 287: 39–58. doi:10.1016/S0304-3975(02)00135-4.
- ^ 1 2 3 Design: Dirks, Robert M.; Lin, Milo; Winfree, Erik & Pierce, Niles A. (15 February 2004). "Paradigms for computational nucleic acid design". Nucleic Acids Research 32 (4): 1392–1403. doi:10.1093/nar/gkh291. PMC 390280. PMID 14990744.
- ^ Methods: Ellington, Andrew; Pollard, Jr., Jack D. (1 May 2001). "Synthesis and purification of oligonucleotides". Current Protocols in Molecular Biology. doi:10.1002/0471142727.mb0211s42. ISBN 0471142727.
- ^ Methods: Ellington, A.; Pollard, Jr., J. D. (1 May 2001). "Purification of oligonucleotides using denaturing polyacrylamide gel electrophoresis". Current Protocols in Molecular Biology. doi:10.1002/0471142727.mb0212s42. ISBN 0471142727.
- ^ Methods: Gallagher, S. R.; Desjardins, P. (1 July 2011). "Quantitation of nucleic acids and proteins". Current Protocols Essential Laboratory Techniques. doi:10.1002/9780470089941.et0202s5. ISBN 0470089938.
- ^ Methods: Chory, Joanne; Pollard, Jr., Jack D. (1 May 2001). "Separation of small DNA fragments by conventional gel electrophoresis". Current Protocols in Molecular Biology. doi:10.1002/0471142727.mb0207s47. ISBN 0471142727.
- ^ Methods: Walter, N. G. (1 February 2003). "Probing RNA structural dynamics and function by fluorescence resonance energy transfer (FRET)". Current Protocols in Nucleic Acid Chemistry. doi:10.1002/0471142700.nc1110s11. ISBN 0471142700.
- ^ Methods: Lin, C.; Ke, Y.; Chhabra, R.; Sharma, J.; Liu, Y.; Yan, H. (2011). "Synthesis and Characterization of Self-Assembled DNA Nanostructures". In Zuccheri, G. and Samor?, B. DNA Nanotechnology: Methods and Protocols. Methods in Molecular Biology. 749. pp. 1–11. doi:10.1007/978-1-61779-142-0_1. ISBN 978-1-61779-141-3.
- ^ Methods: Bloomfield, Victor A.; Crothers, Donald M., Tinoco, Jr., Ignacio (2000). Nucleic acids: structures, properties, and functions. Sausalito, Calif: University Science Books. pp. 84–86, 396–407. ISBN 0-935702-49-0.
- ^ 1 2 3 4 Design: Feldkamp, Udo; Niemeyer, Christof M. (13 March 2006). "Rational design of DNA nanoarchitectures". Angewandte Chemie International Edition 45 (12): 1856–1876. doi:10.1002/anie.200502358. PMID 16470892.
- ^ Overview: Lin, Chenxiang; Liu, Yan; Rinker, Sherri; Yan, Hao (11 August 2006). "DNA tile based self-assembly: building complex nanoarchitectures". ChemPhysChem 7 (8): 1641–1647. doi:10.1002/cphc.200600260. PMID 16832805.
- ^ 1 2 3 Kinetic assembly: Yin, Peng; Choi, Harry M. T.; Calvert, Colby R.; Pierce, Niles A. (17 January 2008). "Programming biomolecular self-assembly pathways". Nature 451 (7176): 318–322. Bibcode 2008Natur.451..318Y. doi:10.1038/nature06451. PMID 18202654.
- ^ DX arrays: Winfree, Erik; Liu, Furong; Wenzler, Lisa A.; Seeman, Nadrian C. (6 August 1998). "Design and self-assembly of two-dimensional DNA crystals". Nature 394 (6693): 529–544. Bibcode 1998Natur.394..539W. doi:10.1038/28998. PMID 9707114.
- ^ DX arrays: Liu, Furong; Sha, Ruojie; Seeman, Nadrian C. (10 February 1999). "Modifying the surface features of two-dimensional DNA crystals". Journal of the American Chemical Society 121 (5): 917–922. doi:10.1021/ja982824a.
- ^ Other arrays: Mao, Chengde; Sun, Weiqiong; Seeman, Nadrian C. (16 June 1999). "Designed two-dimensional DNA Holliday junction arrays visualized by atomic force microscopy". Journal of the American Chemical Society 121 (23): 5437–5443. doi:10.1021/ja9900398.
- ^ Other arrays: Constantinou, Pamela E.; Wang, Tong; Kopatsch, Jens; Israel, Lisa B.; Zhang, Xiaoping; Ding, Baoquan; Sherman, William B.; Wang, Xing; Zheng, Jianping; Sha, Ruojie; Seeman, Nadrian C. (21 September 2006). "Double cohesion in structural DNA nanotechnology". Organic and Biomolecular Chemistry 4 (18): 3414–3419. doi:10.1039/b605212f. PMID 17036134.
- ^ Other arrays: Mathieu, Frederick; Liao, Shiping; Kopatsch, Jens; Wang, Tong; Mao, Chengde; Seeman, Nadrian C. (April 2005). "Six-helix bundles designed from DNA". Nano Letters 5 (4): 661–665. Bibcode 2005NanoL...5..661M. doi:10.1021/nl050084f. PMID 15826105.
- ^ 1 2 Algorithmic self-assembly: Rothemund, Paul W. K.; Papadakis, Nick; Winfree, Erik (December 2004). "Algorithmic self-assembly of DNA Sierpinski triangles". PLoS Biology 2 (12): 2041–2053. doi:10.1371/journal.pbio.0020424. PMC 534809. PMID 15583715.
- ^ Algorithmic self-assembly: Barish, Robert D.; Rothemund, Paul W. K.; Winfree, Erik (December 2005). "Two computational primitives for algorithmic self-assembly: copying and counting". Nano Letters 5 (12): 2586–2592. Bibcode 2005NanoL...5.2586B. doi:10.1021/nl052038l. PMID 16351220.
- ^ Other arrays: Strong, Michael (March 2004). "Protein Nanomachines". PLoS Biology 2 (3): e73. doi:10.1371/journal.pbio.0020073. PMC 368168. PMID 15024422.
- ^ Yan, Hao; Park, Sung Ha; Finkelstein, Gleb; Reif, John H.; Labean, Thomas H. (26 September 2003). "DNA-templated self-assembly of protein arrays and highly conductive nanowires". Science 301 (5641): 1882–1884. doi:10.1126/science.1089389. PMID 14512621.
- ^ DNA nanotubes: Rothemund, Paul W. K.; Ekani-Nkodo, Axel; Papadakis, Nick; Kumar, Ashish; Fygenson, Deborah Kuchnir & Winfree, Erik (22 December 2004). "Design and Characterization of Programmable DNA Nanotubes". Journal of the American Chemical Society 126 (50): 16344–16352. doi:10.1021/ja044319l. PMID 15600335.
- ^ Three-dimensional arrays: Zheng, Jianping; Birktoft, Jens J.; Chen, Yi; Wang, Tong; Sha, Ruojie; Constantinou, Pamela E.; Ginell, Stephan L.; Mao, Chengde; Seeman, Nadrian C. (3 September 2009). "From molecular to macroscopic via the rational design of a self-assembled 3D DNA crystal". Nature 461 (7260): 74–77. Bibcode 2009Natur.461...74Z. doi:10.1038/nature08274. PMC 2764300. PMID 19727196.
- ^ DNA polyhedra: Zhang, Yuwen; Seeman, Nadrian C. (1 March 1994). "Construction of a DNA-truncated octahedron". Journal of the American Chemical Society 116 (5): 1661–1669. doi:10.1021/ja00084a006.
- ^ DNA polyhedra: Shih, William M.; Quispe, Joel D.; Joyce, Gerald F. (12 February 2004). "A 1.7-kilobase single-stranded DNA that folds into a nanoscale octahedron". Nature 427 (6975): 618–621. doi:10.1038/nature02307. PMID 14961116.
- ^ 1 2 DNA boxes: Andersen, Ebbe S.; Dong, Mingdong; Nielsen, Morten M.; Jahn, Kasper; Subramani, Ramesh; Mamdouh, Wael; Golas, Monika M.; Sander, Bjoern; Stark, Holger (7 May 2009). "Self-assembly of a nanoscale DNA box with a controllable lid". Nature 459 (7243): 73–76. Bibcode 2009Natur.459...73A. doi:10.1038/nature07971. PMID 19424153.
- ^ DNA boxes: Ke, Yonggang; Sharma, Jaswinder; Liu, Minghui; Jahn, Kasper; Liu, Yan; Yan, Hao (10 June 2009). "Scaffolded DNA origami of a DNA tetrahedron molecular container". Nano Letters 9 (6): 2445–2447. Bibcode 2009NanoL...9.2445K. doi:10.1021/nl901165f. PMID 19419184.
- ^ Nanoarchitecture: Zheng, Jiwen; Constantinou, Pamela E.; Micheel, Christine; Alivisatos, A. Paul; Kiehl, Richard A.; Seeman Nadrian C. (July 2006). "2D Nanoparticle Arrays Show the Organizational Power of Robust DNA Motifs". Nano Letters 6 (7): 1502–1504. Bibcode 2006NanoL...6.1502Z. doi:10.1021/nl060994c. PMID 16834438.
- ^ Nanoarchitecture: Park, Sung Ha; Pistol, Constantin; Ahn, Sang Jung; Reif, John H.; Lebeck, Alvin R.; Dwyer, Chris; LaBean, Thomas H. (October 2006). "Finite-size, fully addressable DNA tile lattices formed by hierarchical assembly procedures". Angewandte Chemie 118 (40): 749–753. doi:10.1002/ange.200690141.
- ^ Nanoarchitecture: Cohen, Justin D.; Sadowski, John P.; Dervan, Peter B. (22 October 2007). "Addressing single molecules on DNA nanostructures". Angewandte Chemie International Edition 46 (42): 7956–7959. doi:10.1002/anie.200702767. PMID 17763481.
- ^ Nanoarchitecture: Maune, Hareem T.; Han, Si-Ping; Barish, Robert D.; Bockrath, Marc; Goddard, III, William A.; Rothemund, Paul W. K.; Winfree, Erik (January 2009). "Self-assembly of carbon nanotubes into two-dimensional geometries using DNA origami templates". Nature Nanotechnology 5 (1): 61–66. Bibcode 2010NatNa...5...61M. doi:10.1038/nnano.2009.311. PMID 19898497.
- ^ Nanoarchitecture: Liu, Jianfei; Geng, Yanli; Pound, Elisabeth; Gyawali, Shailendra; Ashton, Jeffrey R.; Hickey, John; Woolley, Adam T.; Harb, John N. (22 March 2011). "Metallization of branched DNA origami for nanoelectronic circuit fabrication". ACS Nano 5 (3): 2240–2247. doi:10.1021/nn1035075. PMID 21323323.
- ^ Nanoarchitecture: Deng, Zhaoxiang; Mao, Chengde (6 August 2004). "Molecular lithography with DNA nanostructures". Angewandte Chemie International Edition 43 (31): 4068. doi:10.1002/anie.200460257.
- ^ DNA machines: Mao, Chengde; Sun, Weiqiong; Shen, Zhiyong; Seeman, Nadrian C. (14 January 1999). "A DNA nanomechanical device based on the B-Z transition". Nature 397 (6715): 144–146. Bibcode 1999Natur.397..144M. doi:10.1038/16437. PMID 9923675.
- ^ DNA machines: Yurke, Bernard; Turberfield, Andrew J.; Mills, Allen P., Jr; Simmel, Friedrich C.; Neumann, Jennifer L. (10 August 2000). "A DNA-fuelled molecular machine made of DNA". Nature 406 (6796): 605–609. Bibcode 2000Natur.406..605Y. doi:10.1038/35020524. PMID 10949296.
- ^ DNA machines: Yan, Hao; Zhang, Xiaoping; Shen, Zhiyong; Seeman, Nadrian C. (3 January 2002). "A robust DNA mechanical device controlled by hybridization topology". Nature 415 (6867): 62–65. Bibcode 2002Natur.415...62Y. doi:10.1038/415062a. PMID 11780115.
- ^ DNA machines: Feng, Liping; Park, Sung Ha; Reif, John H.; Yan, Hao (22 September 2003). "A two-state DNA lattice switched by DNA nanoactuator". Angewandte Chemie 115 (36): 4478. doi:10.1002/ange.200351818.
- ^ DNA machines: Goodman, Russell P.; Heilemann, Mike; Doose, S?ren; Erben, Cristoph M.; Kapanidis, Achillefs N.; Turberfield, Andrew J. (February 2008). "Reconfigurable, braced, three-dimensional DNA nanostructures". Nature Nanotechnology 3 (2): 93–96. doi:10.1038/nnano.2008.3. PMID 18654468.
- ^ Applications: Douglas, Shawn M.; Bachelet, Ido; Church, George M. (17 February 2012). "A logic-gated nanorobot for targeted transport of molecular payloads". Science 335 (6070): 831–834. doi:10.1126/science.1214081.
- ^ DNA walkers: Shin, Jong-Shik; Pierce, Niles A. (8 September 2004). "A synthetic DNA walker for molecular transport". Journal of the American Chemical Society 126 (35): 10834–10835. doi:10.1021/ja047543j. PMID 15339155.
- ^ DNA walkers: Sherman, William B.; Seeman, Nadrian C. (July 2004). "A precisely controlled DNA biped walking device". Nano Letters 4 (7): 1203–1207. Bibcode 2004NanoL...4.1203S. doi:10.1021/nl049527q.
- ^ DNA walkers: Tian, Ye; He, Yu; Chen, Yi; Yin, Peng; Mao, Chengde (11 July 2005). "A DNAzyme that walks processively and autonomously along a one-dimensional track". Angewandte Chemie 117 (28): 4429–4432. doi:10.1002/ange.200500703.
- ^ DNA walkers: Bath, Jonathan; Green, Simon J.; Turberfield, Andrew J. (11 July 2005). "A free-running DNA motor powered by a nicking enzyme". Angewandte Chemie International Edition 44 (28): 4358–4361. doi:10.1002/anie.200501262.
- ^ Functional DNA walkers: Lund, Kyle; Manzo, Anthony J.; Dabby, Nadine; Michelotti, Nicole; Johnson-Buck, Alexander; Nangreave, Jeanette; Taylor, Steven; Pei, Renjun; Stojanovic, Milan N.; Walter Nils G.; Winfree, Erik; Yan, Hao (13 May 2010). "Molecular robots guided by prescriptive landscapes". Nature 465 (7295): 206–210. Bibcode 2010Natur.465..206L. doi:10.1038/nature09012. PMC 2907518. PMID 20463735.
- ^ Functional DNA walkers: He, Yu; Liu, David R. (November 2010). "Autonomous multistep organic synthesis in a single isothermal solution mediated by a DNA walker". Nature Nanotechnology 5 (11): 778–782. Bibcode 2010NatNa...5..778H. doi:10.1038/nnano.2010.190. PMC 2974042. PMID 20935654.
- ^ Strand displacement cascades: Seelig, Georg; Soloveichik, David; Zhang, David Y.; Winfree, Erik (8 December 2006). "Enzyme-free nucleic acid logic circuits". Science 314 (5805): 1585–1588. doi:10.1126/science.1132493. PMID 17158324.
- ^ Strand displacement cascades: Qian, Lulu; Winfree, Erik (3 June 2011). "Scaling up digital circuit computation with DNA strand displacement cascades". Science 332 (6034): 1196–1201. doi:10.1126/science.1200520. PMID 21636773.
- ^ Applications: Rietman, Edward A. (2001). Molecular engineering of nanosystems. Springer. pp. 209–212. ISBN 978-0-387-98988-4. Retrieved 17 April 2011.
- ^ Applications: Jungmann, Ralf; Renner, Stephan; Simmel, Friedrich C. (March 2008). "From DNA nanotechnology to synthetic biology". HFSP journal 2 (2): 99–109. doi:10.2976/1.2896331. PMC 2645571. PMID 19404476.